Développement de la bioinformatique appliquée au séquençage haut débit aux multi-omiques

Développement de la bioinformatique appliquée au séquençage haut débit aux multi-omiques
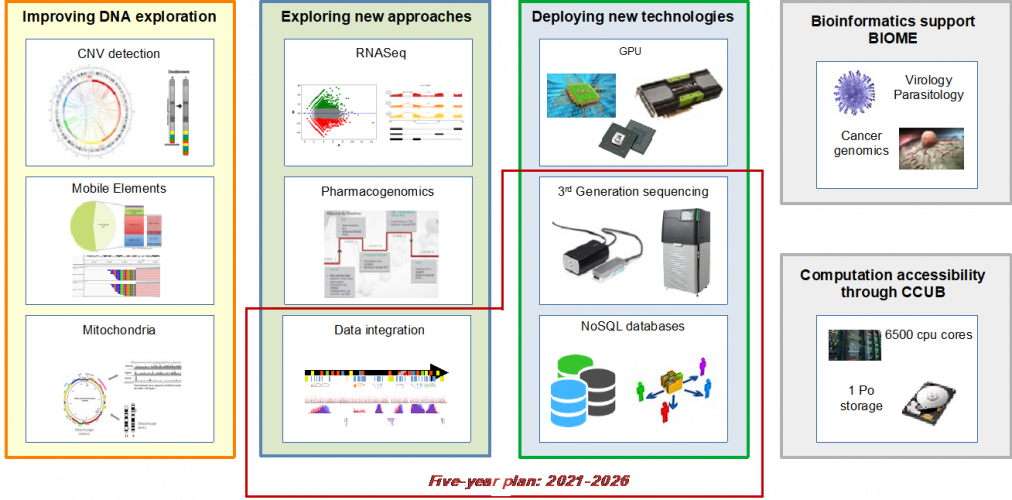
Ce développement s’effectue par le déploiement de processeurs graphiques (GPU) qui constituent une technologie actuellement très peu exploitée d'accélération des temps de calcul des analyses bioinformatiques. Certaines études ont démontré que les calculs réalisés par GPU pouvaient voir leur temps d’exécution être divisés par 10 pour certaines applications bioinformatiques. Les très rares outils existants ne répondent pas aux besoins et à l'évolution actuelle du SHD, le développement de nouveaux outils bioinformatiques utilisant ce type d'architecture constituera donc une avancée majeure dans la bioinformatique moderne appliquée à la génomique humaine, induisant ainsi une forte diminution du temps de réponse diagnostique lié à ces analyses.
Des pipelines dédiés à l’analyse des données de GS short-reads (srGS) et de RNAseq sont également développés, avec plusieurs programmes d’intégration des données (OMIXCARE, INTEGRA et MultiOmixCare).
Par ailleurs, l’évaluation des technologies de séquençage haut débit de 3ème génération (Chromium, 10X, Oxford Nanopore, PacBio) pour l’identification des variations de structure est également un axe de développement majeur.